Dna Double Helix的发现史及测序技术的发展史梳理
2021-09-06
森言森语
测序是日常实验中最为频繁的环节之一,但对于测序原理和技术发展现状往往容易忽略。今天就来梳理一下测序技术的更迭及发展现状。
背景介绍
想要深入了解测序技术,就不得不提DNA双螺旋结构的发现史。
DNA即脱氧核糖核酸,是染色体的主要化学成分和遗传信息的主要载体,其分子结构是由两条核苷酸链组成的双螺旋形。DNA的发现值得人们关注。
1869年,瑞士医生、生化学家米歇尔(J.F.Miescher,1844-1895)在分析细胞的化学组成时,从附近外科诊所的废物箱中捡来满是脓液的绷带,而后用硫酸钠稀溶液冲洗绷带,使细胞保持完好并与脓液中的其他成分分开,得到了很多白血球细胞。然后,他用酸溶解了包围在白血球外面的大部分物质而得到了细胞核,再用稀碱处理细胞核,又得到一种含磷量很高的物质,这种物质引起了他的兴趣,因为这种物质从未有过报道,为此他把位于细胞核中由磷酸产生的酸性基因,一种大分子组成的物质称为“核素”(nuclein)。
1872年又从鲑鱼的精子细胞核中,发现了大量类似的酸性物质,随后有人在多种组织细胞中也发现了这类物质的存在。因为这类物质都是从细胞核中提取出来的,而且都具有酸性,因此,1898年,奥尔特曼首次建议用“核酸”这种名词代替“核素”这个名词。。
米歇尔的德国导师塞勒也从酵母菌中提取出了“核素”。他把酵母中提取出来的“核素”称为“酵母核素”,而米歇尔发现的“核素”由于很容易从动物的胸腺中取得,所以称为“胸腺核素”。
1879年,塞勒的另一名高足、德国生物化学家科塞尔(Kossel,A.1853-1927 )开始系统研究“核素”的结构。他用水解“核素”的办法,经过十多年的研究,从“酵母核素”和“胸腺核素”中,除得到两种嘌呤和两种嘧啶物质外,还发现“核素”中存在碳水化合物。
到20世纪初,科塞尔和他的学生琼斯(Johnew,W.1865-1935 )、列文(Levene,P.A.1896-1940 )已搞清楚核酸的化学成分及其最简单的基本结构。证实它是由四种不同的碱基,即腺嘌呤(A)、鸟嘌呤(G)、胸腺嘧啶(T)和胞嘧啶(C)及核糖、磷酸等组成。其最简单的单体结构是碱基-核糖-磷酸构成的核苷酸。科塞尔也因此获得1910年诺贝尔医学生理学奖。
1929年,俄裔美国生物化学家列文(P.A.leven,1869-1940)发现核酸可分为核糖核酸(RNA)与脱氧核糖核酸(DNA)。
1928和1944年,英国细菌学家格里菲斯(F.Griffith,1877-1941)和美国细菌学家艾弗里(Oswald Averyy,1877-1955)先后通过肺炎双球菌的转化实验证明DNA具有传递遗传信息的功能。
1953年,美国生物学家沃森(Jim Watson,1928- )和英国生物物理学家克里克(Francis Crick,1916-2004),在英国女生物学家富兰克琳(R.Franklin,1920-1958)和英国生物物理学家威尔金斯(M.Wilkins,1916-2004)对DNA晶体所作的X光衍射分析的基础上,根据DNA分子碱基配对原则,构建出了DNA分子的双螺旋结构模型。双螺旋结构显示出DNA分子在细胞分裂时能够被精确复制,解释了其在遗传和进化中的作用。同时,沃森和克里克还预言了遗传信息的复制、传递和表达传递过程是从DNA → RNA→蛋白质,被称为“中心法则”。不久,这一设想被其他科学家的发现所证实。
关于这一段历史,我们可以接着往下了解。
1920年,Rosalind Franklin生于伦敦一个富有的犹太人家庭,15岁就立志要当科学家,但父亲并不支持她这样做。她早年毕业于剑桥大学,专业是物理化学。1945年,获得博士学位之后,她前往法国学习X射线衍射技术。
1951年,她回到英国。那时人们已经知道了DNA是遗传物质,但是对于DNA的结构,以及它如何在生命活动中发挥作用的机制还不甚了解。在DNA重要的基础上,对DNA结构的解析就十分行重要。就在这时,Franklin加入了研究DNA结构的行列,然而当时的环境相当不友善。她开始负责实验室的DNA项目时,有好几个月没有人干活。同事Wilkins不喜欢她进入自己的研究领域,但他在研究上却又离不开她。
他把她看做搞技术的副手,她却认为自己与Wilkins地位同等,两人的私交恶劣到几乎不讲话。当时的剑桥,对女科学家的歧视处处存在,女性甚至不被准许在高级休息室里用午餐。她们无形中被排除在科学家间的联系网络之外,而这种联系对了解新的研究动态、交换新理念、触发灵感极为重要。
Franklin在法国学习的X射线衍射技术在研究中派上了用场。X射线是波长非常短的电磁波。医生通常用它来透视人体,而物理学家用它来分析晶体的结构。当X射线穿过晶体之后,会形成样一种特定的明暗交替的衍射图形。不同的晶体产生不同的衍射图样,仔细分析这种图形人们就能知道组成晶体的原子是如何排列的。Franklin成功地拍摄了DNA晶体的X射线衍射照片。
Watson和Crick也在剑桥大学卡文迪许实验室进行DNA结构的研究,Watson在美国本来是从事噬菌体遗传学研究的,他们希望通过噬菌体来搞清楚基因如何控制生物的遗传。导师派他出国学习并没有生硬地规定课题,甚至他从一个国家的实验室到另一个国家的实验室也能得到导师的支持或谅解。当他听了Wilkins的学术报告,看到DNA的X射线衍射图片后,认定一旦搞清DNA的结构,就能了解基因如何起作用。这是他集中精力从事DNA结构研究的最大契机。于是他不等批准,就决定先斩后奏从丹麦去伦敦学习X射线衍射技术了。至于Crick,他是个不拘小节又相当狂妄的聪明人,不太受“老板”Bragg欢迎,甚至一度有可能被炒鱿鱼。但是,当因为学术问题引起的误会消除后,老板照样关心他的工作,在那篇划时代的论文写成后,Bragg认真修改并热情地写信向《Nature》推荐。这种现象在一个以学术为重的研究机构应该是正常的。人际关系对研究事业的干扰是轻微的。
1953年的Watson和Crick都是名不见经传的小人物,37岁的Crick连博士学位还没有得到。受到前人的影响,他们原来按照3股螺旋的思路进行了很长时间的工作,可是既构建不出合理模型,也遭到结晶学专家Franklin的强烈反对,结果使工作陷于僵局。在发现正确的双股螺旋结构前2个月,他们看到蛋白质结构权威Pauling一篇即将发表的关于DNA结构的论文,Pauling错误地确定为3股螺旋。Watson在认真考虑并向同事们请教后,决然地否定了权威的结论。正是在否定权威之后,他们加快了工作,在不到两个月内终于取得了后来震惊世界的成果。
1952年,奥地利裔美国生物化学家查伽夫(E.chargaff,1905-2002)测定了DNA中4种碱基的含量,发现其中腺嘌呤与胸腺嘧啶的数量相等,鸟嘌呤与胞嘧啶的数量相等。这使沃森、克里克立即想到4种碱基之间存在着两两对应的关系,形成了腺嘌呤与胸腺嘧啶配对、鸟嘌呤与胞嘧啶配对的概念。
在科学界经常遇到的是年轻人对权威无原则的屈服,甚至Watson在开始知道鲍林提出的是三螺旋模型的一刹那,也曾后悔几个月前放弃了自己按三螺旋思路进行的工作。不过他们没有从此打住,而是为了赢得时间,加快了工作。因为他们相信这是智者Pauling千虑之一失,很快本人就会发现错误并迅速得出正确结论。Wilkins在Franklin不知情的情况下给他们看了那张照片。根据照片,整日焦虑于DNA结构发现的Watson和Crick立即领悟到了现在已经成为众所周知的事实——两条以磷酸为骨架的链相互缠绕形成了双螺旋结构,氢键把它们连结在一起。他们在1953年5月25日出版的英国《Nature》杂志上报告了这一发现。双螺旋结构显示出DNA分子在细胞分裂时能够自我复制,完善地解释了生命体要繁衍后代,物种要保持稳定,细胞内必须有遗传属性和复制能力的机制。这是生物学的一座里程碑,分子生物学时代的开端,怎样评价其重要性都不过分。
在1953年2月底,33岁的Franklin已经在日记中写道,DNA具有两条链的结构。这时她已经确认这个生物分子具有两种形式,链外面有磷酸根基团。1953年3月17日,当Franklin将研究结果整理成文打算发表时,发现Watson和Crick破解DNA结构的消息已经出现在新闻简报中。4月2日,Watson、Crick和Wilkins的文章送交《Nature》杂志,4月25日发表,接着他们在5月30日的《Nature》杂志上又发表了“DNA的遗传学意义”一文,更加详细地阐述了DNA双螺旋模型在功能上的意义。1953年初, Watson和Crick构建出DNA分子双螺旋结构模型,而此时Franklin对这一进展并不知情。她更不知道的是,Watson和Crick曾看过她拍摄的能验证DNA双螺旋结构的X射线晶体衍射照片,并由此获得了重要启发。
Franklin的贡献是毋庸置疑的:她分辨出了DNA的两种构型,并成功地拍摄了它的X射线衍射照片。Watson和Crick未经她的许可使用了这张照片,但她并不在意,反而为他们的发现感到高兴,还在《Nature》杂志上发表了一篇证实DNA双螺旋结构的文章。
Watson在1968年出版的《双螺旋》一书中坦承,“Franklin没有直接给我们她的数据”。而Crick在很多年后也承认,“她离真相只有两步”。目前,科技界对Franklin的工作给予较高评价,对Wilkins是否有资格分享发现DNA双螺旋结构的殊荣存在很大争论。
1962年,当Watson、Crick和Wilkins共同分享诺贝尔奖时,Franklin已经因长期接触放射性物质而患卵巢癌英年早逝。
这个故事的结局还是很伤感的。
英国剑桥大学的卡文迪许实验室,一直坚持这样的规定:每天下午六点整,老资格的研究人员来到实验室,宣布时间已到,要求每个人停止工作。如果谁不遵守,他们便引用Rutherford的话加以劝导。Rutherford说过:“谁未能完成六点前必须完成的工作,也就没有必要拖延下去,倒是希望各位马上回家,好好想想今天做的工作,好好思考明天要做的工作。”那是一天深夜,Rutherford披着外衣,又来到实验室检查,惊奇地发现有人还在做实验。由于低头,又十分专心,那学生没发现Rutherford站在他的身后。Rutherford轻声地问道:“你上午干什么?”学生回头一看,是Rutherford,他马上站起来,小心地回答:“做实验。”Rutherford又问:“那么,下午呢?”学生回答:“做实验。”Rutherford提高了声调,再问:“晚上呢?”学生以为老师在表扬他,得意地回答:“还是做实验。”Rutherford极为严肃地问:“你整天做实验,还有时间去认真思考吗?!”那学生低下了头。临走,Rutherford告诫他:“别忘了思考!”从此,卡文迪许实验室的人记住了Rutherford的忠告:“别忘了思考!”
不管怎样,DNA及其双螺旋结构的发现,把分子生物学推向了全世界,“生命之谜”也伴随着对遗传密码的破译和测序技术的发展而逐渐拉开序幕。
不论如何,值得回顾这一段历史。
DNA是遗传物质,DNA包含了生命的全部秘密,人们已经迫不及待想要把DNA看的一清二楚,而解密的关键在于测序。下面就来回顾一下测序技术的发展。
一代测序技术
DNA测序技术,即获得目的DNA片段碱基排列顺序的技术,获得目的DNA片段的序列是进一步进行分子生物学研究和基因改造的基础。
1970年,中国工程院外籍院士、生物化学家吴瑞首先发明了DNA测序方法以及其他一些DNA克隆技术。
1971年,吴瑞将引物延伸(primer extension)用于DNA测序,成为Sanger法测序之重要一步,引物延伸也用于其他两项诺贝尔奖的工作中:Kary Mullis的PCR,和Michael Smith的定点突变。开创了DNA测序技术的先河。
1955年,Sanger将胰岛素的氨基酸序列完整地定序出来,同时证明蛋白质具有明确构造。他利用自己新发现的Sanger试剂(Sanger’s reagent),也就是2,4-二硝基氟苯(2,4-dinitrofluorobenzene)将胰岛素降解成小片段,并与专门水解蛋白质的胰蛋白酶混合在一起。再将一部分混合物的样本置放于滤纸的一面,并利用一种色层分析方法来做进一步的实验,首先他将一种溶剂从单一方向通过滤纸,同时又让电流以相反向通过。
由于不同的蛋白质片段有不同的溶解度与电荷,因此在电泳后,这些片段最后会各自停留在不同的位置,产生特定的图案。Sanger将此图案称为“fingerprints”;不同的蛋白质拥有不同的图案,成为可供辨识且可重现的特征。之后fingerprints又将小片段重新组合成氨基酸长链,进而推导出完整的胰岛素结构。因此得出结论,认为胰岛素具有特定的氨基酸序列。这项研究使他单独获得了1958年的诺贝尔化学奖。
1975年,Sanger和Coulson发明了“Plus and Minus”(俗称“加减法”)测定DNA序列
1977年Maxam and Gilbert等人建立了化学降解法测序。同年,Sanger引入ddNTP(双脱氧核苷三磷酸),发明了著名的双脱氧链终止法,又称“Sanger法”,于1980年与另外两名科学家共享诺贝尔化学奖。双脱氧链终止法有效控制了化学降解法中化学毒素和同位素的危害。
1977年,Walter Gilbert和Frederick Sanger发明了第一台测序仪,并应用其测定了第一个基因组序列,噬菌体X174,全长5375个碱基。自此,人类获得了窥探生命遗传差异本质的能力,并以此为开端步入基因组学时代。
测序原理
利用一种DNA聚合酶来延伸结合在待定序列模板上的引物。直到掺入一种链终止核苷酸为止。
每一次序列测定由一套四个单独的反应构成,每个反应含有所有四种脱氧核苷酸三磷酸(dNTP),并混入限量的一种不同的双脱氧核苷三磷酸(ddNTP)。由于ddNTP缺乏延伸所需要的3-OH基团,使延长的寡聚核苷酸选择性地在G、A、T或C处终止。终止点由反应中相应的双脱氧而定。
每一种dNTPs和ddNTPs的相对浓度可以调整,使反应得到一组长几百至几千碱基的链终止产物。它们具有共同的起始点,但终止在不同的的核苷酸上,可通过高分辨率变性凝胶电泳分离大小不同的片段,凝胶处理后可用X-光胶片放射自显影或非同位素标记进行检测。
DNA的复制需要:DNA聚合酶,双链DNA模板,带有3'-OH末端的单链寡核苷酸引物,4种dNTP(dATP、dGTP、dTTP和dCTP)。聚合酶用模板作指导,不断地将dNTP加到引物的3'-OH末端,使引物延伸,合成出新的互补DNA链。如果加入一种特殊核苷酸,双脱氧核苷三磷酸(ddNTP),因它在脱氧核糖的3’位置缺少一个羟基,故不能同后续的dNTP形成磷酸二酯键。
如,存在ddCTP、dCTP和三种其他的dNTP(其中一种为α-32P标记)的情况下,将引物、模板和DNA聚合酶一起保温,即可形成一种全部具有相同的5'-引物端和以ddC残基为3’端结尾的一系列长短不一片段的混合物。经变性聚丙烯酰胺凝胶电泳分离制得的放射性自显影区带图谱将为新合成的不同长度的DNA链中C的分布提供准确信息,从而将全部C的位置确定下来。
类似的方法,在ddATP、ddGTP和ddTTP存在的条件下,可同时制得分别以ddA、ddG和ddT残基为3‘端结尾的三组长短不一的片段。将制得的四组混合物平行地点加在变性聚丙烯酰胺凝胶电泳板上进行电泳,每组制品中的各个组分将按其链长的不同得到分离,制得相应的放射性自显影图谱。从所得图谱即可直接读得DNA的碱基序列。与DNA复制不同的是sanger测序中的引物是单引物或者是单链。
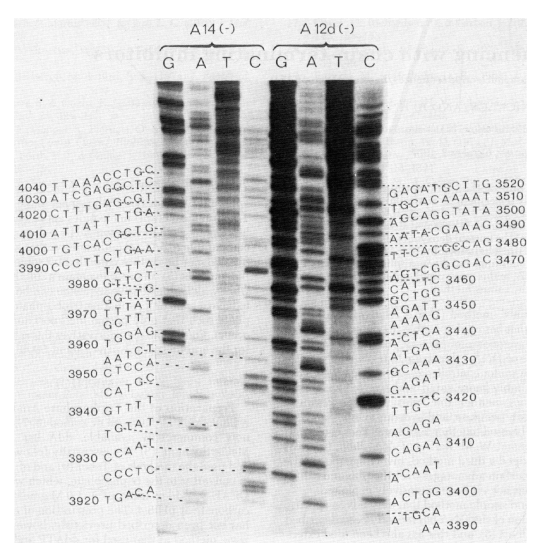 引自文章,但看起来不很清楚。
引自文章,但看起来不很清楚。
 引自网络,从这张放射自显影图中可直观的读出序列。也就很好理解其原理了。由此也可以推断出模板链的序列。
引自网络,从这张放射自显影图中可直观的读出序列。也就很好理解其原理了。由此也可以推断出模板链的序列。
Sanger测序流程
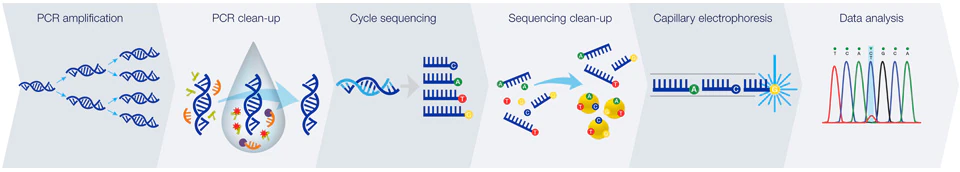
- PCR扩增片段或提取质粒
- 分离纯化待测序的质粒或PCR扩增产物
- DNA模板定量
- 进行测序PCR反应
- 测序反应后纯化
- 毛细管电泳
- 数据分析
优点
- 准确度高于二三代测序;
- 每个反应可以得到700-1000 bp的序列,序列长度高于二代测序;
劣势
- 一个反应只能得到一条序列,测序通量底;
- 测序成本很高。
早在2001年完成的人类基因组框图,采用的就是Sanger测序法,而且因其准确率高,至今仍然被广泛使用,也被称为基因检测的金标准。也正是这一特点,而被应用于临床。
二代测序技术
第一代测序技术的主要特点是测序读长可达1000bp,准确性高达99.999%,但其测序成本高,通量低等方面的缺点,严重影响了其真正大规模的应用。因而第一代测序技术并不是最理想的测序方法。经过不断的技术开发和改进,以Roche公司的454技术、illumina公司的Solexa,Hiseq技术和ABI公司的Solid技术为标记的第二代测序技术诞生了。第二代测序技术大大降低了测序成本的同时,还大幅提高了测序速度,并且保持了高准确性,以前完成一个人类基因组的测序需要3年时间,而使用二代测序技术则仅仅需要1周,但在序列读长方面比起第一代测序技术则要短很多。
第二代 DNA 测序技术又称高通量测序技术(high—throughput sequencing,HTS),还被称为下一代测序(Next-generation sequencing,NGS),深度测序(deep sequencing)是基于PCR和基因芯片发展而来的DNA测序技术。
2005年454公司的 Genome Sequencer 20 System,2006年illumina公司的Solexa和2007年ABI公司的SOLiD标志着第二代测序技术的诞生。 之后陆续在2006年推出454 FLX系统、2010年推出illumina HiSeq系统和同年推出的基于半导体芯片测序技术的Ion PGM已经陆续成为当前基因测序的中坚力量。
一代测序为合成终止测序,而二代测序开创性的引入了可逆终止末端,从而实现边合成边测序(Sequencing by Synthesis)。二代测序在DNA复制过程中通过捕捉新添加的碱基所携带的特殊标记(一般为荧光分子标记)来确定DNA的序列。
测序原理
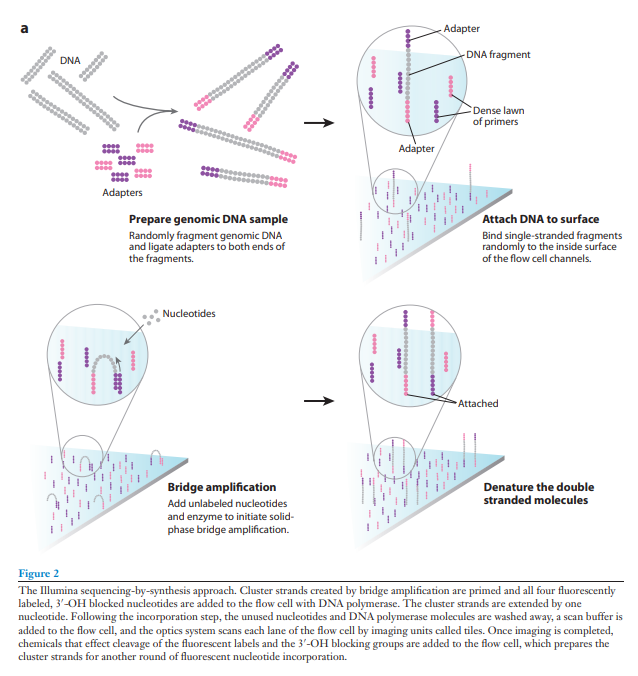
简要的原理如图,在整理过程中看到有位知乎大佬写了一个详细便于理解的原理。遂引用如下:
基本原理 基于可逆终止的,荧光标记dNTP,做边合成边测序 分为三步:
- Sample Prep
- Cluster Generation
- Sequencing
- Data Analysis
1、Sample Prep 通过不同实验方法得到的样品,需要先提取样本基因组中的DNA,用超声波将其随机打断。然后使用酶将两端补平,使用Klenow 酶在3‘ 端加一个 A 碱基(用于连接接头序列)。为了后续扩增,测序分析,需要为这些DNA片段添加特定的接头序列。接头序列是已知的,大概有三种:sequencing binding site(绿) index(红,黄) 流动池引物互补的序列(蓝,紫) 添加完接头序列后的DNA片段集合叫DNA文库 (DNA library),这样就完成了样品准备工作。
2、Cluster Generation 成簇是DNA片段被扩增的过程,该过程在流动池 (Flowcell) 中完成。它是一片带有8条通道(lanes)的玻璃载玻,每个通道内表面附有两种DNA引物。首先,引物会与样品中的DNA片段的接头序列互补配对,固定在通道表面
通过聚合酶生成杂交片段的互补片段,然后加入NaOH碱溶液后,双链分子变性,原始模板链(左边的链)被流动池中的液体洗去
加入中性液体用于中和碱溶液,剩下的单链拷贝链另一端的接头就会与通道表面的引物结合,形成单链桥。
同样的,在聚合酶参与下,生成互补链,最终形成双链桥
通过变性,DNA分子线性化,变为两个单链拷贝
它们又分别与自己配对的引物结合
重复这个循环,同时形成数百万的簇。在这个过程中,所有的DNA片段都会被克隆扩增。
桥式扩增后,反向链会被切断洗去,仅留下正向链。为防止特异性结合重新形成单链桥,3‘端被封锁
3、Sequencing 首先,在Flowcell中加入荧光标记的dNTP和酶,由引物起始开始合成子链。但是dNTP存在 3’端叠氮基会阻碍子链延伸,这使得每个循环只能测得一个碱基。合成完一个碱基后, Flowcell 通入液体洗掉多余的dNTP和酶,使用显微镜的激光扫描特征荧光信号。
荧光发射波长与信号强度一起决定了碱基的读出,所有的DNA片段的一个碱基会被同时读取。在大规模并行的过程中,机器读取的图像类似下面这样
加入化学试剂将叠氮基团与荧光基团切除,然后 Flowcell 再通入荧光标记的dNTP和酶,由引物起始开始合成一个碱基。不断重复这个过程,完成第一次读取。 由于测序仪每次测序时的通量比较大,所以每次测得的序列可能不止一个样本。为了去区分每个样本及正负链,科学家构建DNA文库时,在接头序列加入了的不同 index(或 barcode)来区分来源。 首先,在完成第一次读取后,复制出的链会被洗去
index 片段引物被引入并与模板杂交,完成序列读取后被洗去。这样读取到的序列与开始时已知的index比对后就可以给测得的序列贴上标签,方便后续分析。
Paired-end测序已经是现在的主流,它提高了测序长度的同时,又可以为结构变异分析提供新方法。要完成双末端测序,首先要将模板链3’去保护,模板折叠,index片段引入
在聚合酶参与下形成双链桥
然后变性,恢复为单链。注意,这次是将正向链切除并洗去,只留下反向链
反向链以测序引物为起始,与正向链类似,经过多个循环后完成读取。
4、Data Analysis 测序完成后会产生数百万个 reads,基于在样品准备时构建的 index 分类来自不同样本的序列。对于每个样品来说,具有相似延伸的碱基被聚在一起。正向和反向read配对生成连续序列。这些序列通过与参考基因组匹配后,实现完整序列的构建。
三代测序技术
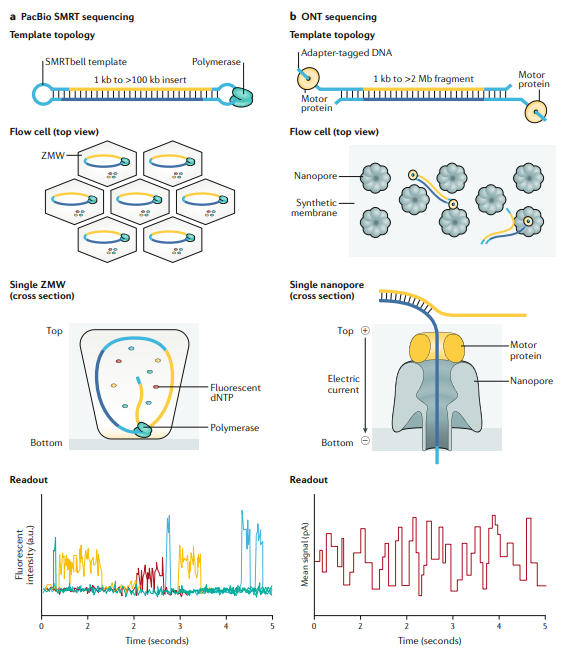
第三代测序技术是指单分子测序技术。DNA测序时,不需要经过PCR扩增,实现了对每一条DNA分子的单独测序。第三代测序技术也叫从头测序技术,即单分子实时DNA测序。
第三代测序技术原理主要分为两大技术阵营: 第一大阵营是单分子荧光测序,代表性的技术为美国螺旋生物(Helicos)的SMS技术和美国太平洋生物(Pacific Bioscience)的SMRT技术。脱氧核苷酸用荧光标记,显微镜可以实时记录荧光的强度变化。当荧光标记的脱氧核苷酸被掺入DNA链的时候,它的荧光就同时能在DNA链上探测到。当它与DNA链形成化学键的时候,它的荧光基团就被DNA聚合酶切除,荧光消失。这种荧光标记的脱氧核苷酸不会影响DNA聚合酶的活性,并且在荧光被切除之后,合成的DNA链和天然的DNA链完全一样。
第二大阵营为纳米孔测序,代表性的公司为英国牛津纳米孔公司。新型纳米孔测序法(nanopore sequencing)是采用电泳技术,借助电泳驱动单个分子逐一通过纳米孔 来实现测序的。由于纳米孔的直径非常细小,仅允许单个核酸聚合物通过,而ATCG单个碱基的带电性质不一样,通过电信号的差异就能检测出通过的碱基类别,从而实现测序。
测序原理
PacBio的 Single Molecule Real-Time(SMRT)Sequencing
SMRT技术应用ZMW(zero-mode waveguide,零模式波导)
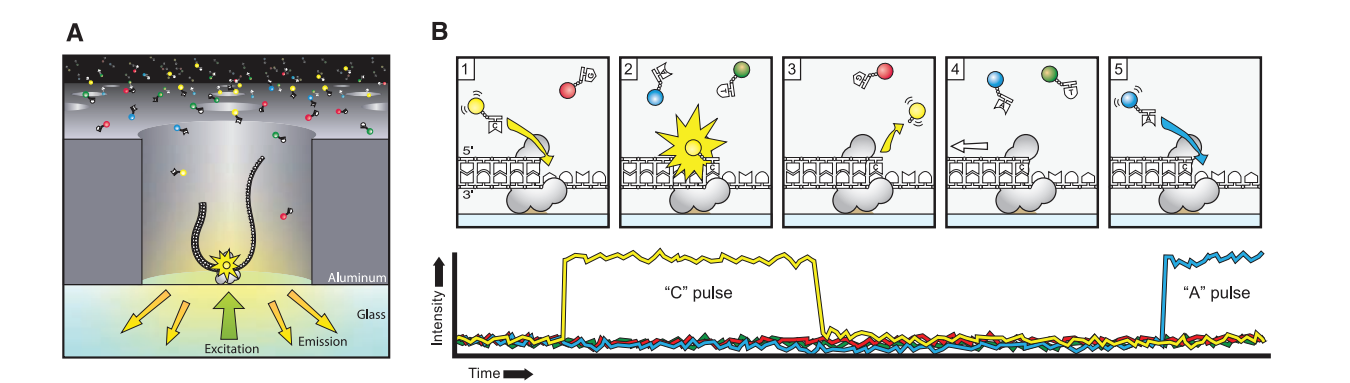 引自文章
ZMW是一个直径为几十纳米的小孔,它阻止可见的激光(波长大约为600 nm)完全透过ZMW。激光在进入ZMW后迅速衰减,因此,只有底部30 nm被照亮。在每个ZMW中,单个DNA聚合酶分子利用专利技术锚定在底部玻璃的表面。随后核苷酸涌入ZMW中,并在阵列表面扩散。当聚合酶检测到正确的核苷酸时,便将其掺入新生链中,这个过程需要几毫秒,而单纯的扩散只需要几微秒。这种时间差使掺入的核苷酸产生了很高的信号强度,类似于脉冲信号。因此,ZMW有能力在荧光标记核苷酸的背景下检测单个掺入事件。
引自文章
ZMW是一个直径为几十纳米的小孔,它阻止可见的激光(波长大约为600 nm)完全透过ZMW。激光在进入ZMW后迅速衰减,因此,只有底部30 nm被照亮。在每个ZMW中,单个DNA聚合酶分子利用专利技术锚定在底部玻璃的表面。随后核苷酸涌入ZMW中,并在阵列表面扩散。当聚合酶检测到正确的核苷酸时,便将其掺入新生链中,这个过程需要几毫秒,而单纯的扩散只需要几微秒。这种时间差使掺入的核苷酸产生了很高的信号强度,类似于脉冲信号。因此,ZMW有能力在荧光标记核苷酸的背景下检测单个掺入事件。
为了观察掺入事件,使用了一种纳米光子结构,即零模波导(ZMW),相对于共焦荧光显微镜,它可以将观察体积减少三个数量级以上。尽管DNA聚合酶需要相对较高的标记dNTP浓度(0.1-10mm),但这种限制水平可以实现单荧光团检测,以实现快速、准确和过程性合成。与全内反射显微镜的约3到300摩尔相比,该范围产生的平均分子占位在直径为100 nm的ZMW的约0.01到1个分子之间。
测序是在专利的SMRT Cell中开展的,每个Cell中有一个阵列,上面有15万个ZMW。每个ZMW都能够包含一个DNA聚合酶及一条不同的DNA样品链。每次运行能够平行检测大约75,000个单分子测序反应。
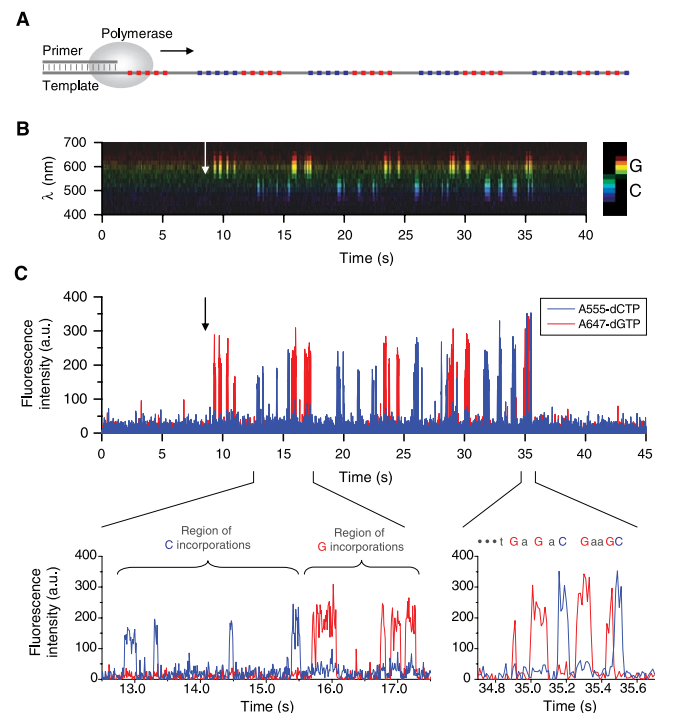 引自文章
引自文章
 引自文章
引自文章
the Advantages of SMRT Sequencing
- Long Reads
With reads tens of kilobases in length you can readily assemble complete genomes and sequence full-length transcripts. - High Accuracy
Sequencing free of systematic error achieves >99.999% consensus accuracy. Explore the benefits of highly accurate long-read sequencing. - Uniform Coverage
No bias based on GC content means you can sequence through regions inaccessible to other technologies. - Single-Molecule Resolution
Capturing sequence data from native DNA or RNA molecules enables highly accurate long reads with >99.9% single-molecule accuracy. - Epigenetics
With no PCR amplification step, base modifications are directly detected during sequencing.
Nanopore DNA sequencing
参考文献
WATSON JD, CRICK FH. Molecular structure of nucleic acids; a structure for deoxyribose nucleic acid. Nature. 1953;171(4356):737-738. doi:10.1038/171737a0
Sanger F, Coulson AR, Barrell BG, Smith AJ, Roe BA. Cloning in single-stranded bacteriophage as an aid to rapid DNA sequencing. J Mol Biol. 1980;143(2):161-178. doi:10.1016/0022-2836(80)90196-5
Logsdon GA, Vollger MR, Eichler EE. Long-read human genome sequencing and its applications. Nat Rev Genet. 2020;21(10):597-614. doi:10.1038/s41576-020-0236-x
Zhao S, Ye Z, Stanton R. Misuse of RPKM or TPM normalization when comparing across samples and sequencing protocols. RNA. 2020;26(8):903-909. doi:10.1261/rna.074922.120
Mardis ER. Next-generation DNA sequencing methods. Annu Rev Genomics Hum Genet. 2008;9:387-402. doi:10.1146/annurev.genom.9.081307.164359
Eid J, Fehr A, Gray J, et al. Real-time DNA sequencing from single polymerase molecules. Science. 2009;323(5910):133-138. doi:10.1126/science.1162986
McCombie WR, McPherson JD, Mardis ER. Next-Generation Sequencing Technologies. Cold Spring Harb Perspect Med. 2019;9(11):a036798. Published 2019 Nov 1. doi:10.1101/cshperspect.a036798
Sanger F, Nicklen S, Coulson AR. DNA sequencing with chain-terminating inhibitors. Proc Natl Acad Sci U S A. 1977;74(12):5463-5467. doi:10.1073/pnas.74.12.5463
Maxam AM, Gilbert W. A new method for sequencing DNA. Proc Natl Acad Sci U S A. 1977;74(2):560-564. doi:10.1073/pnas.74.2.560
参考网络资源
https://www.youtube.com/watch?v=E9-Rm5AoZGw https://nanoporetech.com/ https://www.pacb.com/ http://www.biotrainee.com/jmzeng/book/basic/sequencing.html#sanger https://blog.csdn.net/u011262253/article/details/102525491 https://zhuanlan.zhihu.com/p/91913739 https://baike.baidu.com/item/%E7%AC%AC%E4%B8%89%E4%BB%A3%E6%B5%8B%E5%BA%8F%E6%8A%80%E6%9C%AF/2349961?fr=aladdin